相同組換えは遺伝学でもバイオテクノロジーでも重要な生命現象です。しかし専門用語がたくさん出てくるので理解しにくい現象です。今回は相同組換えについてできるだけわかりやすく解説していきたいと思います。
相同組換えのしくみ
そもそも相同組換えとは?
相同組換えは、切断された二本鎖DNAを配列が類似した(場合によっては同一の)二本鎖DNAを用いて修復する過程の中でそれぞれの二本鎖DNAがもつ遺伝情報の組み合わせが変わる現象です。
DNAの二本鎖が切れてしまうと遺伝子の情報が分断されたり、切られたDNAが分解されたりしてしまいます。そうならないために切れたDNAをつなぎ直します。
ただし、この時完全に元どおりのDNA配列に修復されず、DNA修復の最後に絡まったDNAの鎖を解くときに、修復した二本鎖DNAと修復に使った二本鎖DNAの構成が大きく入れ替わることもあります。
ではDNA修復の過程では何が起きているのでしょうか?
DNAレベルの分子メカニズム
DNAレベルで切断されたDNAの修復過程を見ていきましょう。4つの修復過程を紹介しますが、以下の2つのステップは共通しています。
- DNAの二本鎖切断
- 切れ端部分の5'末端を分解
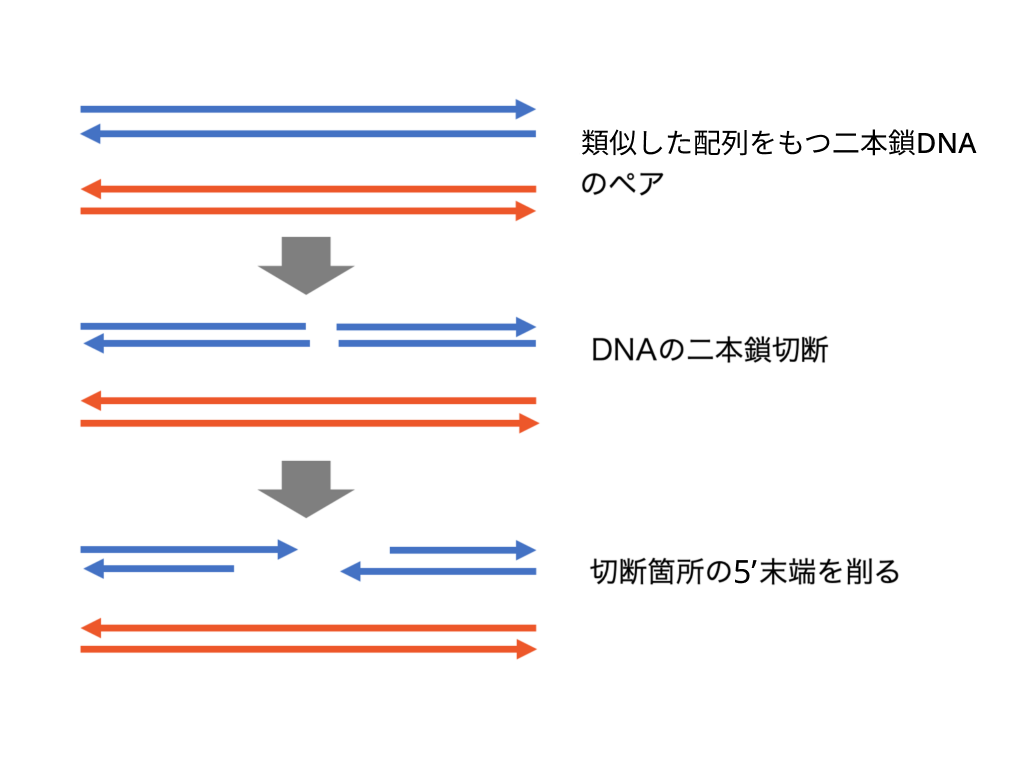
二本鎖切断を受けたDNAはまず切断箇所の5’末端を分解します。そうすることで切断箇所の3’末端だけが一本鎖DNAの状態になります(この状態のDNAを3'オーバーハングといいます)。その後に、二本鎖切断を受けたDNAと類似した配列をもつDNAを鋳型にして、3'オーバーハングからDNAを合成し直します。
相同組換えに関わるDNAの修復過程には以下の4つの方法があります。
- Double strand break repair (DSBR)
- synthesis-dependent strand-annealing (SDSA)
- Single strand annealing (SSA)
- Break-induced replication (BIR)
一番重要なのは一番上のDSBRです。
順番に見ていきましょう。
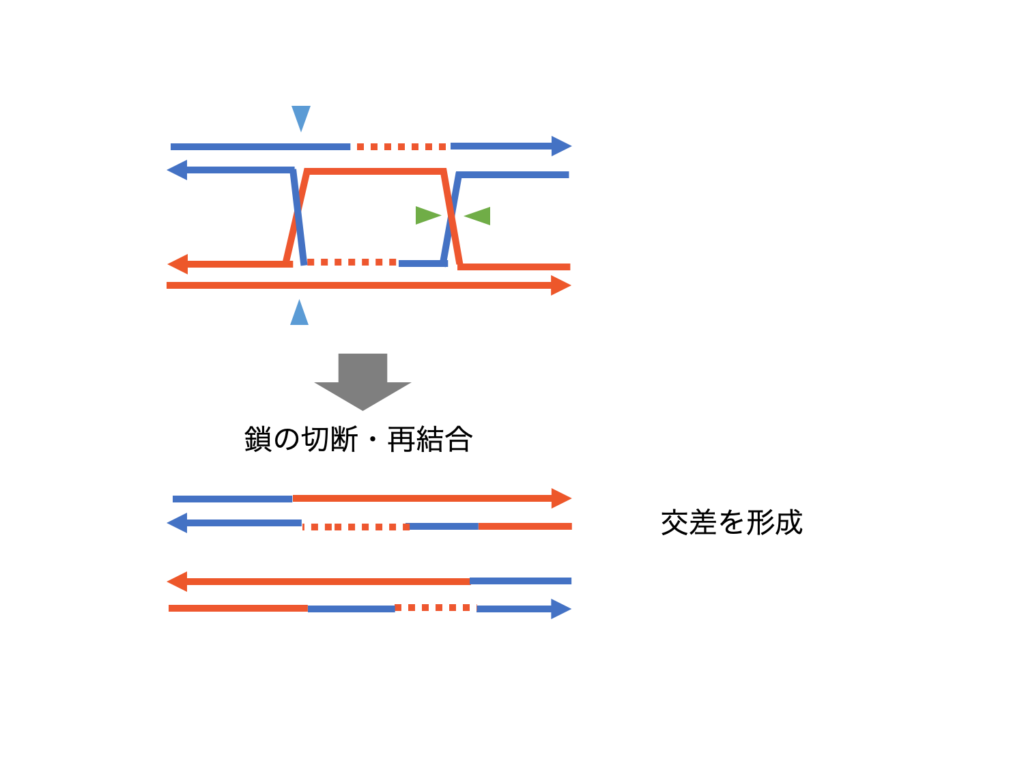
Double strand break repair (DSBR)
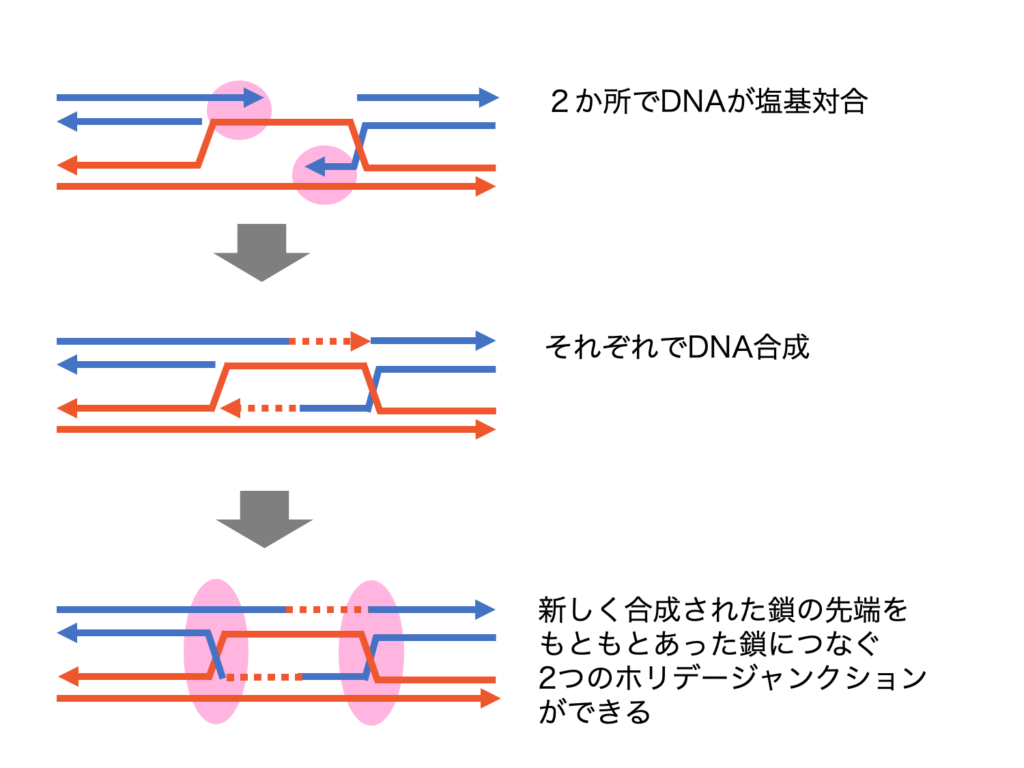
DSBRは、DNAの構成を大きく変える可能性がある相同組換えです。2つの3'オーバーハングをそれぞれを鋳型になるDNAと対合させて切れてしまったDNAを合成し直します。
このDNA合成は、ホリデージャンクション(Holiday junction)と呼ばれる、合成したDNAが絡み合ったような構造を作ります。
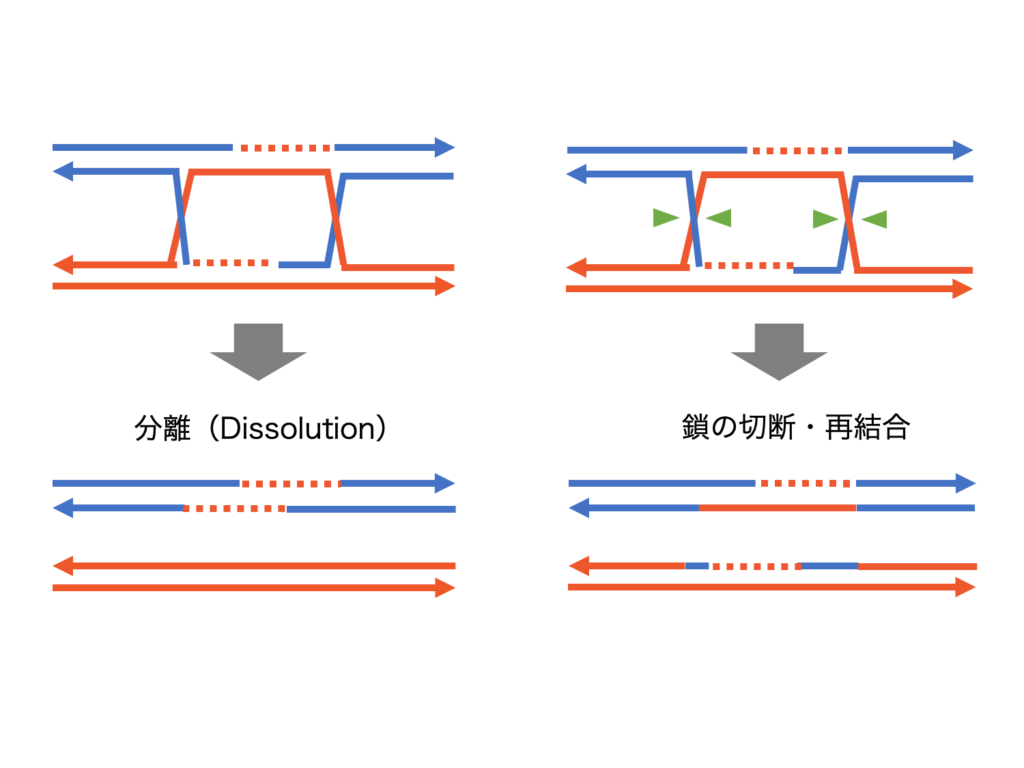
ホリデージャンクションの構造は複雑なので、下の図をみてください。このホリデージャンクションの解消の仕方によってDNAの構成は大きく変わることがあります。DNA切断で戻す方法と塩基対合し直す分離(Dissolution)という方法があります。
ホリデージャンクションを解消するためにDNA切断が必要ですが、その切断の仕方は2パターンあります。図中では水色と緑の矢じりで区別しました。水色で示した部分でDNAを切断して再結合させたときにできるDNAと緑で示した部分でDNAを切断して再結合させたときにできるDNAの構成は変わってきます。
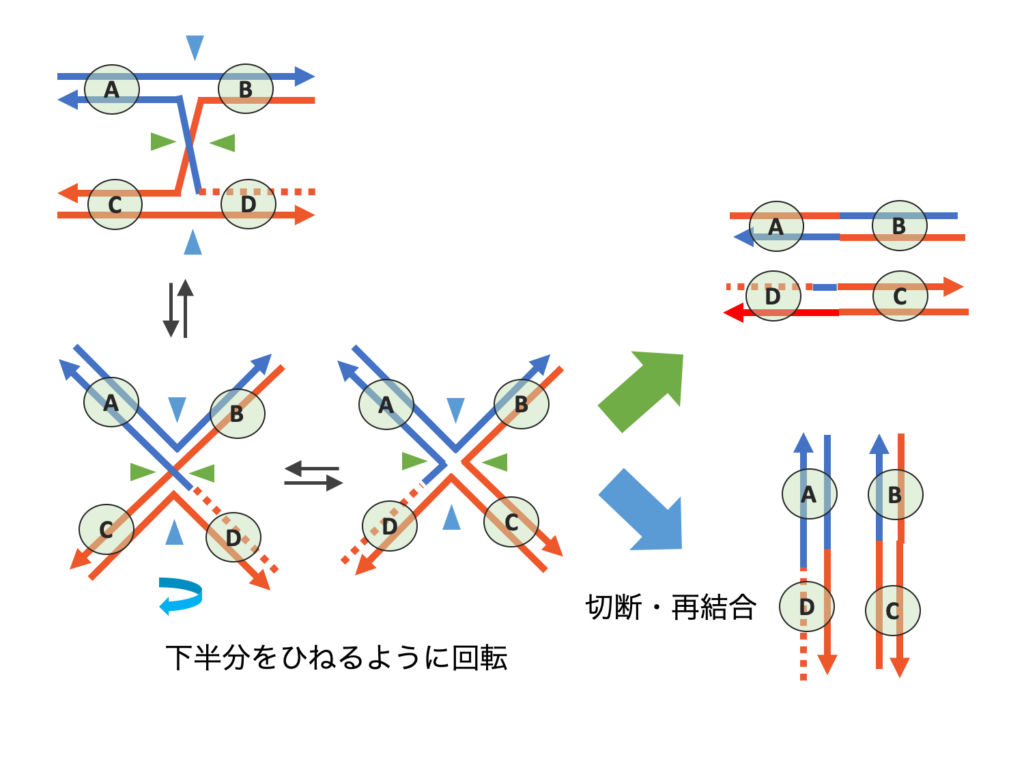
DSBRでは2つのホリデージャンクションが作られます。片方を水色パターン、もう一方を緑パターンで切断すると、DNAはもう一方のDNA(対合していた類似した配列をもつDNA)につながれます。この現象を交差(Cross over)といいます。またホリデージャンクションでのDNA切断をどちらも緑のパターンで行うと交差なしでDNAが解かれます。
Synthesis-dependent strand-annealing (SDSA)
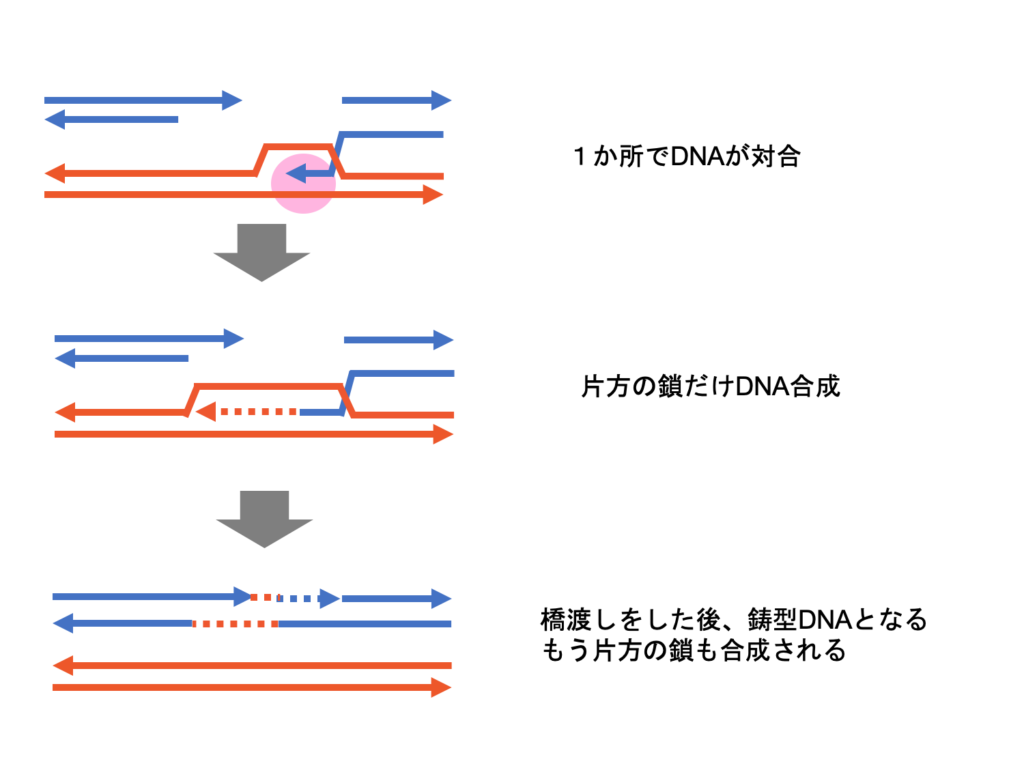
この方法は1つ前のDSBRと違い、一本鎖だけを類似した配列をもつDNAを鋳型にして合成します。もう1つの鎖は類似したDNA配列と対合しません。差し込んだDNAの一本鎖を引き抜いて、その一本鎖DNAで切れ端同士を橋渡しをすることでDNAをつなぎ直し、鋳型をもとにDNAを合成し直します。切れ端同士をつなぎ合わせたあとは類似した配列をもつDNAは必要なくなります
Single strand annealing (SSA)
一番シンプルな修復方法です。切れ端のある反復配列を使って直付けする方法です。他の修復方法と違って、類似した配列を持つDNAを使いません。その代わり切れたDNAにある繰り返し配列を使います。
切れたDNAの末端同士に相同な繰り返し配列がある場合に、紙テープと紙テープをのりでつなげるように、DNAの鎖の末端を同士を対合させます。
マイクロサテライトと呼ばれるような重要な意味はなく反復する配列でDNAの二本鎖切断が起こった場合には、簡単にDNAをつなぎ合わせることができる有効な方法と考えられます。
ただし完全な修復ではなく、切断された場所の近くにある配列(対合した2つの配列の間にあった配列)がなくなってしまうので、DNAの長さは短くなってしまいます。
Break-induced replication (BIR)
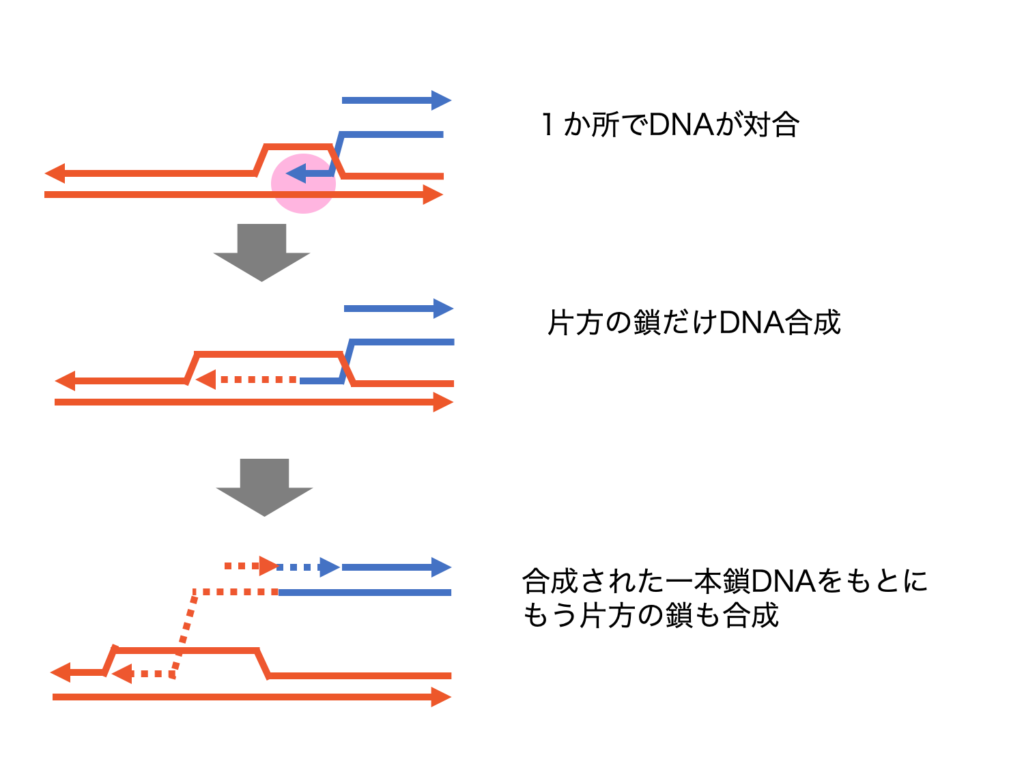
途中で終わっているDNAの続きを類似した配列をもつDNAを鋳型にして合成していく方法です。DNAの片方の鎖だけを類似した配列をもつDNAを鋳型にして合成して、もう一方の鎖はその新しく合成された鎖を鋳型に作っていきます。DNA複製中に複製が止まってしまい、切り離されてしまった中途半端な染色体の修復や染色体の末端にあるテロメアを伸ばす方法にもなります。
相同組換えの役割
一部の配列は類似しているが、全体としては異なる遺伝情報をもつ二本鎖DNA同士(例えば父系染色体と母系染色体)の相同組換えでは、交差を起こすことによって二本鎖DNAが持つ遺伝情報の構成を大きく変えることができます。これは遺伝的多様性を高める働きがあります。また相同組換えはバイオテクノロジーで遺伝子組換えを行うときにも用いられます。DNA修復のときに使う類似したDNA配列の間に外来の遺伝子を入れることで染色体の中にその遺伝子をかきこませることができます。



コメント
コメント失礼します。
とても丁寧で分かりやすいです!
ページ作成時のミスだと思うのですが、SSAの説明イラストがSDSAと同じになっています。SDSAのイラストもあればぜひ見たいです。
昔の記事なのにすみません。よろしくお願いします。
コメントありがとうございます!
ご指摘の通り、SDSAのイラストをSSAにも貼っていました。
申し訳ございません。
SSAのイラストは作成していなかったため、
“Single strand annealing”で画像検索していただけると幸いです。
複数の組換えのメカニズムの違いを分かりやすくまとめていて素晴らしいと思います。ただし間違っている点やあいまいな点があるように思いますので2点コメントします。
まず最初に3’末端を削るとありますが,これは5’末端の間違いだと思います。突出鎖を他のDNA鎖と二本鎖形成させてさらに伸長するのであれば,突出部は3’末端でないといけません。ということは削り込むのは切断部の5’末端ということになる筈です。
もう一点気になったのは,体細胞におけるDNA修復(二本鎖切断修復)の相同組換え修復と,生殖細胞系列で起こる減数分裂第一分裂での相同組換えを混同しているように思える点です。体細胞ではそれぞれの相同染色体は核内で独立に存在し,それぞれのテリトリーを形成しているので空間的に参照可能な程の近さには位置しません。したがってこれらが互いに乗り換えを起こすことはまずありえません。損傷修復の際に鋳型とされるのは,損傷前に複製済みの姉妹染色分体です。姉妹染色分体同士は互いに完全に同じ配列ですし,M期後期に染色体分配が行われるまでは物理的に一体となって染色体を形成していますので,一方が損傷された際に修復の鋳型として参照することが可能です。一方,減数分裂の交差は父方母方それぞれに由来し,細かく見るとところどころ塩基配列も異なる相同染色体間での組換えです。減数分裂第一分裂ではS期に複製された相同染色体同士がさらに対になり,4本の染色分体からなる二価染色体が形成されます。これは体細胞分裂のM期では見られない染色体です。この二価染色体において,「非」姉妹染色分体間(相同染色体間)で組換えが起こるのが相同組換えであり,組換えの前後で染色体の配列をあえて多様化させることに生理的意義があると思います。
以上ご確認下さい。
長文失礼致しました。
コメントありがとうございます。
> まず最初に3’末端を削るとありますが,これは5’末端の間違いだと思います。
申し訳ございません、間違いでしたので修正したしました。
> もう一点気になったのは,体細胞におけるDNA修復(二本鎖切断修復)の相同組換え修復と,生殖細胞系列で起こる減数分裂第一分裂での相同組換えを混同しているように思える点です。
混同しているように読み取れる内容を修正いたしました。
相同な染色体と記載している箇所が多くありましたが、配列が類似している二本鎖DNAとすべきでした。
一般的なDNAの修復方法についての説明の中で、重要なのは類似した配列をもつ二本鎖DNAであることです。この場合、姉妹染色体や外来の二本鎖DNAでもよいため、父系母系の染色体のペアを意味する相同染色体という記述は特定の条件(減数分裂時)のみの説明になってしまうため誤っていました。